Il premio Nobel alla medicina 2024 è il frutto di un sapere condiviso negli anni
9 min letturaPer chi fa ricerca biomedica l'annuncio del premio Nobel per la medicina e fisiologia è sempre un momento atteso con curiosità. Consapevoli però del fatto che il premio va a una, magari due persone, come se avessero fatto tutto loro. Ma, senza nulla togliere alla determinazione, all'intuito, alle capacità di chi viene premiato, la scienza non funziona così.
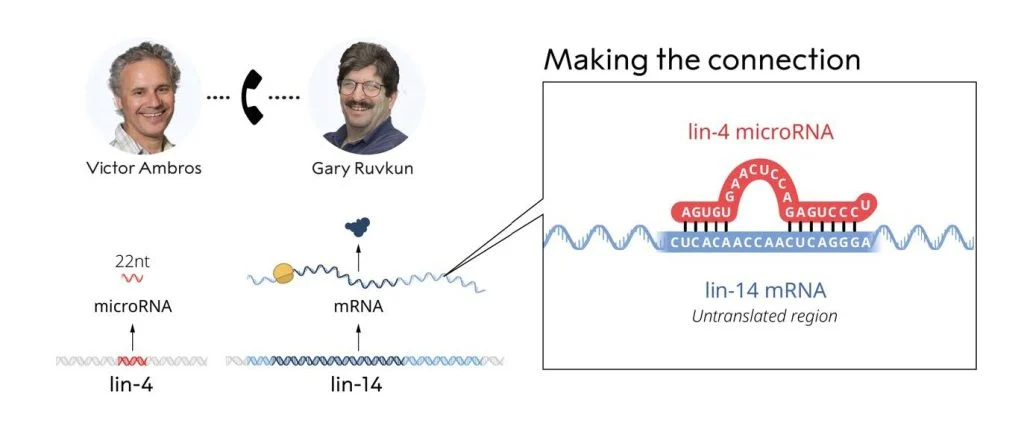
La scienza è cooperazione. Proprio i due ricercatori premiati quest'anno, Victor Ambros e Gary Ruvkun, ce lo dimostrano. Hanno iniziato il loro percorso insieme, poi hanno continuato su vie separate, ma il momento determinante della loro storia è l'11 giugno del 1992 quando i due si sono parlati e hanno messo in condivisione i loro dati (le sequenze di due geni, Lin-4 e Lin-14 appena ottenute dopo anni di lavoro), e solo in quel modo hanno trovato quello che stavano cercando e non avrebbero trovato da soli.
Non è scontato, troppo spesso la sfiducia negli altri e la presunzione di sapere tutto ci fa sprecare ottime possibilità.
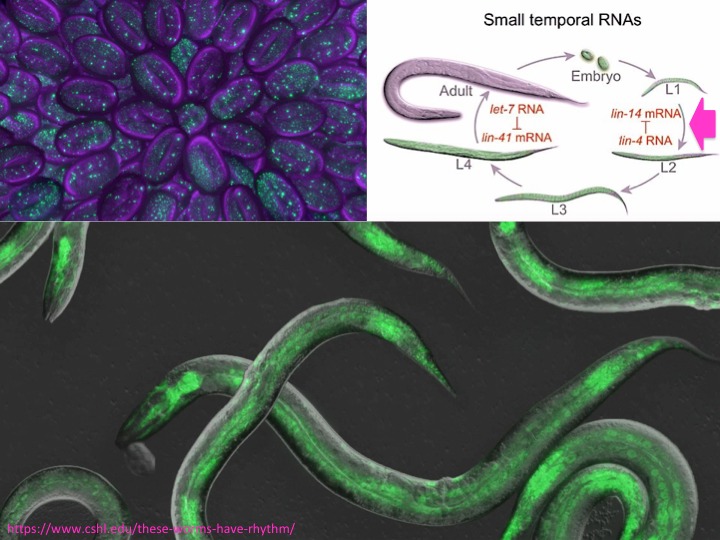
La scienza si costruisce sulla conoscenza condivisa e lasciata a disposizione da altri prima di noi. Ruvkun ed Ambros hanno lavorato su un modello sperimentale, un piccolo nematode chiamato Caenorhabditis elegans, che da quando è diventato una star del laboratorio tutti chiamano solo C. elengans (pron. "si-elegans"). C. elegans era particolarmente adatto per le loro ricerche sullo sviluppo della larva, perché è un organismo semplice, ha un ciclo vitale breve, è facile ottenere mutanti per scoprire a che servono i vari geni coinvolti, ed essendo completamente trasparente è possibile vedere quello che succede dentro senza "romperlo", specialmente se usi marcature fluorescenti, come quelle nelle foto (embrioni ed adulti di C.elegans). Il modello era stato messo a punto negli anni '70 e poi lasciato a disposizione della comunità scientifica da Sydney Brenner, Robert Horvitz e John Sulston. Che nel 2002 avrebbero ricevuto il premio Nobel per le scoperte sulla morte cellulare programmata (apoptosi) fatte proprio in C. elegans.
Anche l'idea che l'attività di un gene potesse essere modulata agendo sul suo RNA messaggero non era nuova. Nella nostra frenesia di mettere le mani nella cellula, come approccio sperimentale si usavano già gli oligonucleotidi antisenso, dei piccoli pezzi di DNA complementari all’mRNA del gene da spegnere. L'ibrido DNA/RNA infatti non veniva tradotto in proteina ma veniva degradato (purtroppo anche gli oligo antisenso venivano degradati in fretta, nonostante la chimica sofisticata su cui si basavano).
Anche il fatto che degli RNA antisenso (complementari al gene di interesse) e a doppio filamento (in fondo simili a quelli che Ruvkun ed Ambros avrebbero scoperto) fossero in grado di silenziare un gene, era già noto, il fenomeno si chiamava RNAi (RNA interference). E per quello nel 2006 Andrew Fire e Craig Mello avrebbero ricevuto il premio Nobel.
Insomma il merito delle grandi scoperte è individuale, ma il lavoro che le ha consentite è collettivo, e spesso non è mirato a quella scoperta in particolare.
Questo implica che l'approccio sempre più meritocratico, che tende sempre più a selezionare poche eccellenze tra cui distribuire la maggior parte di risorse sempre più scarse per tutti gli altri, forse sembra vincente (un caso esemplare di darwinismo sociale applicato alla scienza) ma alle lunghe non avrà effetti positivi.
Ruvkun ed Ambros del resto non lavoravano sull'RNA, l'argomento per cui pochi giorni fa sono stati premiati.
Lavoravano sui meccanismi molecolari che controllano lo sviluppo della larva del C.elegans. In particolare (ora ci vuole un po' di pazienza...) su quei meccanismi che danno il timing corretto al passaggio da uno stadio larvale a quello successivo (da L1 a L2, ci sono 4 stadi larvali prima di diventare un nematode adulto). A molti può sembrare poco eccitante o poco utile, ma la biologia dello sviluppo era molto in voga in quegli anni, ci ha dato informazioni fondamentali, e C.elegans era un ottimo modello perché passa dall'essere una singola cellula ad essere un organismo con strutture funzionali composto da 959 cellule differenziate in un tempo che varia da 3 a 4 giorni, a seconda delle condizioni.
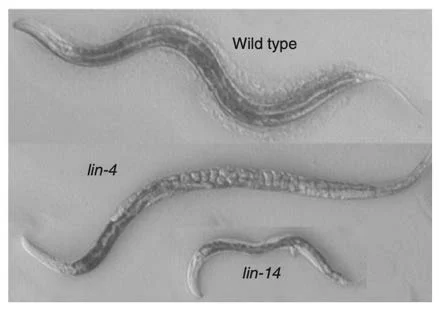
Ruvkun ed Ambros in particolare scoprirono che il passaggio da L1 ad L2 era controllato dall'interazione tra il gene Lin-14 ed il gene Lin-4. Il gene Lin-14 è presente durante tutta la fase precoce L1 ma sparisce improvvisamente al momento del passaggio ad L2, proprio mentre appare Lin-4. Se Lin-4 non c’è, Lin-14 resta attivo e la larva non completa lo sviluppo.
Erano gli anni in cui si scoprivano i fattori di trascrizione, proteine che legano il DNA di un gene a monte della regione codificante e ne regolano la trascrizione (ai tempi credevamo anche che ci fossero attivatori e repressori della trascrizione, poi avremmo capito che era un po' più complicato). Era scontato in quel momento pensare che Lin-4 era un repressore di Lin-14. Solo che Lin-4 non codificava per una proteina in grado di legare il DNA.
A quel punto Ruvkun ed Ambros scoprirono che l'RNA del gene repressore Lin-4, che appariva al momento del passaggio da L1 a L2, quando Lin-14 spariva, si trovava nelle cellule in due forme molto particolari, una lunga circa 60 nucleotidi ed una circa 20 nucleotidi. Che era veramente strano perché gli RNA messaggeri in genere sono lunghi centinaia o migliaia di nucleotidi. Se trovi pezzi corti è perché sono degradati. Solo scambiandosi e confrontando le loro rispettive sequenze, scoprirono che nella parte terminale dell’mRNA di Lin-14 c'erano diverse piccole regioni di omologia con la sequenza di una regione di Lin-4. E questo fu l'inizio di tutto.
Per farla breve (ma solo per chi vuole sapere cosa è stato scoperto), e alla luce di quello che si sarebbe saputo più tardi, avevano scoperto che quello del gene repressore Lin-4 non era un RNA messaggero codificante per una proteina, ma il primo di una famiglia molto grande di RNA che saranno chiamati ncRNA (non codificanti) il cui trascritto originario (pri-miRNA) veniva editato da un complesso proteico nel nucleo che degradava tutto lasciando intatto solo un frammento di circa 60 nucleotidi (pre-miRNA) caratterizzato da una sequenza particolare. Questo frammento infatti era composto da due regioni ripetute, una invertita rispetto all'altra, separate da uno spaziatore. Siccome l'RNA, che è un singolo filamento, tende ad appaiare tutte le regioni con sequenza complementare per raggiungere maggiore stabilità strutturale, questo frammento di 60 nucleotidi creava quello che si chiama uno stem-loop, o anche hair-pin (la forcina per i capelli), ovvero una lunga regione appaiata a doppio filamento con un'ansa terminale a singolo filamento. Il palloncino dei disegnini, guardando la figura si capisce meglio.
L'RNA è una molecola strana e meravigliosa, a differenza del DNA le sue funzioni non sono affidate solo alla sua sequenza nucleotidica, ma anche alla sua struttura 2D e 3D determinata da quella sequenza. Un po' come i Nobel per la chimica di quest’anno ci hanno spiegato che fanno le proteine.
Il frammento di 60 nucleotidi (pre-miRNA) a causa della sua struttura viene riconosciuto da un secondo complesso proteico che lo porta fuori dal nucleo, dove sarà di nuovo editato da un terzo complesso, che ne lascerà solo un piccolo frammento a doppio filamento di 22 nucleotidi.
Ora succede la cosa incredibile: il frammento di RNA da 22 nucleotidi nel citoplasma interagisce con un quarto complesso proteico e uno dei due filamenti viene allontanato e degradato. Ciò che resta di tutto un gene trascritto alla fine è un piccolo frammento di RNA a singolo filamento di 22 nucleotidi, il miRNA maturo. Il miRNA maturo si andrà a pescare nel citoplasma tutti quegli mRNA in cui sono presenti delle regioni di sequenza a cui appaiarsi, come quelle scoperte da Ambros e Ruvkun in Lin-14. Per l'appaiamento serve in genere una regione di almeno sette nucleotidi (seed) e l'appaiamento prevede anche dei nucleotidi non appaiati, che creano una struttura riconoscibile da altre proteine.
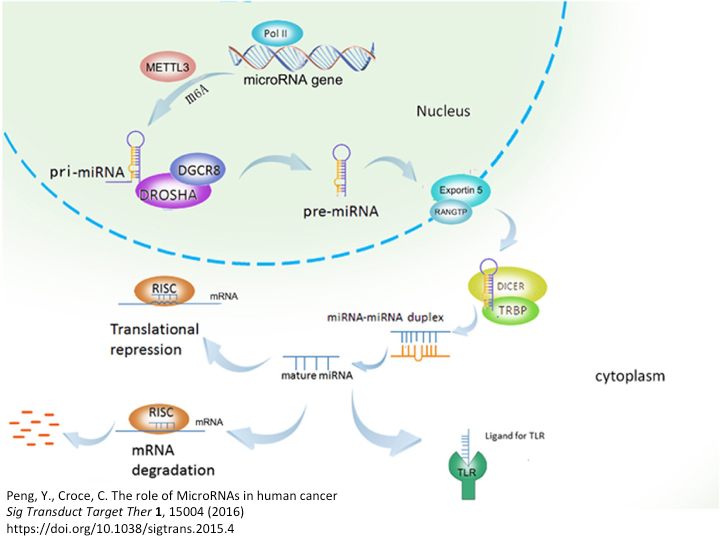
Quello che succede a questo punto è complicato. L’mRNA a cui si è attaccato il miRNA può andare incontro semplicemente alla repressione della sua traduzione in proteina, oppure sarà degradato. In entrambi i casi la proteina del gene represso sparirà in fretta dalla cellula (e con essa la sua funzione) ed il gene risulterà silenziato. Ma non è una regolazione della trascrizione, il gene viene regolarmente trascritto ma contemporaneamente silenziato, un po' come guidare l’auto azionando contemporaneamente l’acceleratore ed il freno. Nel primo caso però (blocco della traduzione) la modulazione è transitoria, nel secondo (degradazione dell’mRNA) è più stabile. Si sta scoprendo che gli RNA messaggeri legati da un miRNA e dal complesso inibitorio collegato (RISC), vengono sequestrati temporaneamente in strutture particolari del citoplasma da cui o saranno rilasciati e rimandati ai ribosomi a far proteine oppure saranno degradati, secondo quello che decide la cellula.
Quindi, anche se con modalità diverse, si tratta sempre di una forma di controllo post-trascrizionale.
La scoperta di tutto questo nella larva di C.elegans fruttò a Ruvkun ed Ambros due pubblicazioni nello stesso numero di Cell del dicembre 1993 (back to back, è prassi quando due gruppi arrivano insieme a risultati simili o collegati) ma al momento non ci fu tanta risonanza, perché sembrava un bel giochino limitato però a un gene di un nematode. Poco dopo però Ruvkun scoprì un secondo microRNA (Let-7) sempre in C.elegans. E nel 2001 si scoprì che Let-7 aveva degli omologhi evolutivamente conservati nel DNA di animali che si erano separati da C.elegans milioni di anni prima. Ed un sistema simile era presente anche nelle piante. Quindi non era un giochino esclusivo di un nematode. Ben presto si capì che era un sistema in grado di controllare in modo coordinato l’attività di molti geni contemporaneamente, diffuso in modo universale nel mondo animale e vegetale. E questo decisamente cambiava tutto.
Ovviamente, se con la scoperta del microRNA Lin-4 si è risolto il problema di chi facesse sparire Lin-14, si è creato un nuovo problema: chi regola Lin-4?
Alla regolazione della trascrizione dei geni codificanti per proteine si era aggiunto un nuovo livello di complessità, la regolazione della trascrizione degli RNA non codificanti, compresi i miRNA. Ormai sappiamo che le regioni codificanti (anche se è un termine sbagliato) per i microRNA possono stare un po' ovunque, all'interno di geni già noti e codificanti per proteine, in regioni di DNA non codificante...E ormai sappiamo che alcuni miRNA e più genericamente molti ncRNA sono coinvolti nei meccanismi epigenetici con cui la cellula controlla la trascrizione dei suoi geni. Sed quis custodiet ipsos custodes? Chi controlla i controllori?
Se poi qualcuno si chiede a cosa sia servito studiare la larva di un nematode, oggi sappiamo che i miRNA sono coinvolti in quasi tutti i processi cellulari (sviluppo, differenziamento, mantenimento funzionale, omeostasi). Sappiamo che uno stesso miRNA può regolare geni diversi, e che un gene può essere regolato da miRNA diversi.
Sappiamo che le cellule mandano in giro per l’organismo microvescicole contenenti i propri miRNA perché arrivino a portare modifiche in altre cellule, anche distanti.
Sappiamo che la trascrizione non regolata di molti miRNA è associata a patologie cardiache, neuronali, al cancro. Allo stesso modo in cui abbiamo gli oncogeni (geni attivi in modo anomalo nel tumore) abbiamo gli oncomiR, miRNA attivi in modo deregolato nel tumore, che andranno per esempio a spegnere geni oncosoppressori…
E se qualcuno si chiede, come ha fatto un giornalista presente alla conferenza stampa dell’annuncio del premio, “ma a che ci serviranno questi miRNA?”, ovviamente qualcuno ha pensato di combattere il nemico con le sue stesse armi, e se il cancro schiera gli oncomiR noi risponderemo con gli antagomir, dei miRNA che vadano a neutralizzare selettivamente i miRNA deregolati. E magari, siccome l’appetito vien mangiando, perché non intervenire con i miRNA o gli antagomiR giusti per ottenere qualche cambiamento mirato? Si però... abbiamo detto che lo steso miRNA può regolare molti geni, e un gene può essere regolato da più miRNA. E poi non c'è una specificità per cellula o tessuto.
La strada è ancora lunga, dunque, ma questo sarà l'argomento di un'altra storia, qui si parlava solo di Lin-4, di Lin-14 e di una larva di un nematode lungo meno di un millimetro.
Immagine in anteprima via nobel prize







